Para propulsar aviones, los motores a reacción necesitan combustibles densos y ricos en energía. Actualmente, casi todo ese combustible proviene del petróleo, ya que las baterías aún no ofrecen la potencia suficiente para la mayoría de los vuelos. Los científicos llevan mucho tiempo soñando con una alternativa sintética: enseñar a los microbios a fermentar material vegetal para producir combustibles para aviones de alto rendimiento. Sin embargo, el diseño de estas "minifábricas" microbianas ha sido tradicionalmente lento y costoso debido a la imprevisibilidad de los sistemas biológicos.
En dos estudios recientes, dos equipos del Instituto Conjunto de Bioenergía (JBEI), gestionado por el Laboratorio Nacional Lawrence Berkeley (Berkeley Lab), han demostrado métodos complementarios para acelerar drásticamente este proceso. Uno combina inteligencia artificial y automatización de laboratorio para probar y refinar rápidamente los diseños genéticos de los microbios productores de biocombustibles. El otro convierte el "mal hábito" de un microbio en una potente herramienta de detección, descubriendo vías ocultas que impulsan la producción.
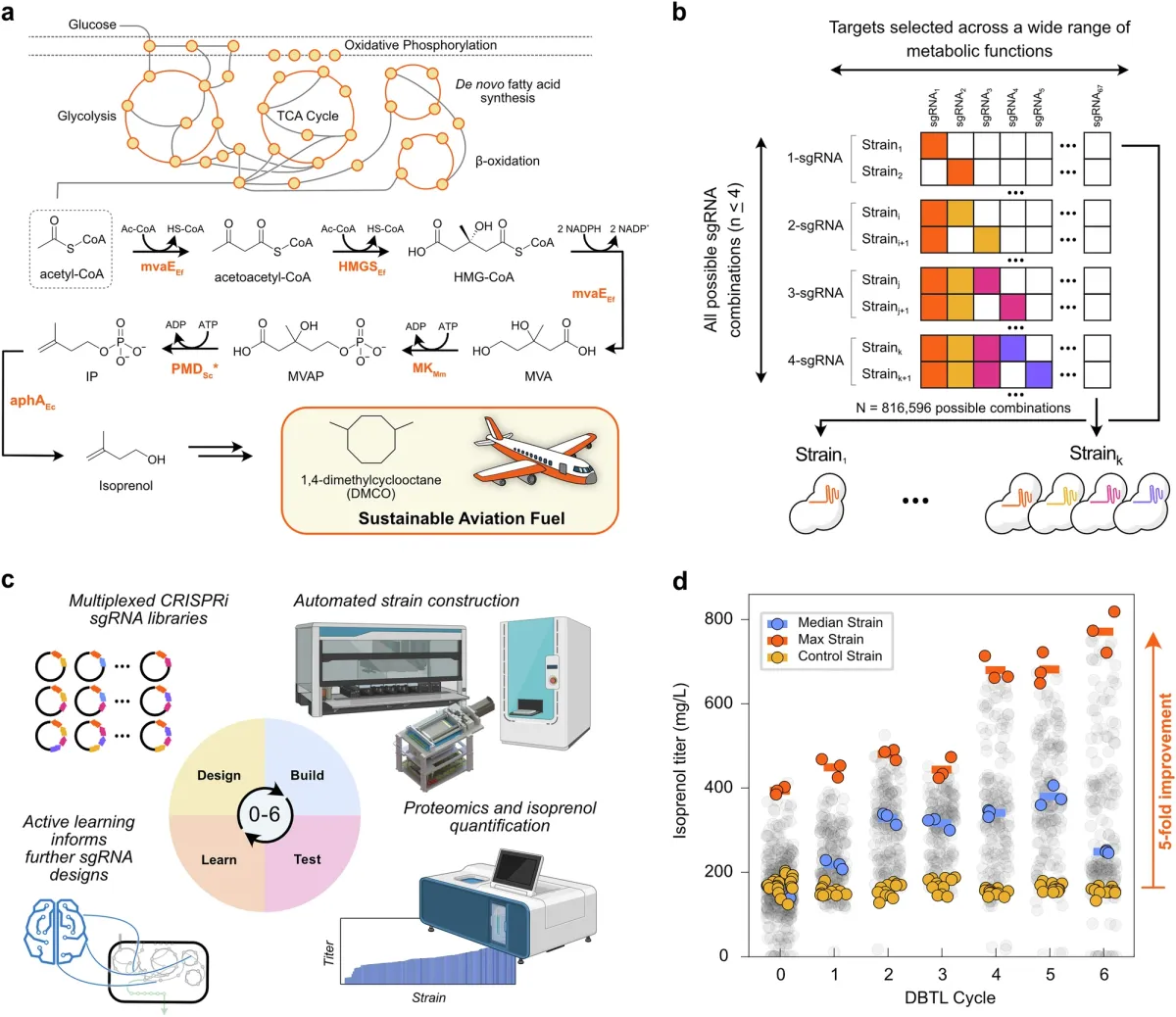
Su objetivo común es el isoprenol, un alcohol transparente y volátil que puede convertirse en DMCO, un combustible para aviones de nueva generación con mayor densidad energética que los combustibles de aviación convencionales actuales. La producción eficiente de isoprenol ha sido un desafío constante en la biología sintética.
Los dos estudios —uno publicado en Nature Communications y el otro en Science Advances— abordan diferentes aspectos de este desafío. El primero utiliza la automatización y el aprendizaje automático para diseñar cepas de Pseudomonas putida que producen cinco veces más isoprenol que antes. El segundo enfoque convierte la capacidad natural de la bacteria para detectar combustibles en una ventaja. Al reconfigurar ese sistema para convertirlo en un biosensor, el equipo pudo analizar rápidamente millones de variantes e identificar cepas que producen hasta 36 veces más isoprenol.
“Se trata de dos estrategias complementarias muy eficaces”, afirmó Thomas Eng, autor principal del estudio sobre biosensores, subdirector de Ingeniería de Huéspedes del JBEI e investigador de la División de Sistemas Biológicos e Ingeniería (BSE) del Laboratorio Berkeley. “Una es la optimización basada en datos; la otra, el descubrimiento. Juntas, nos permiten avanzar mucho más rápido que el método tradicional de ensayo y error”.
Pero quedémosnos sólo con la primera donde se usa la Inteligencia Artificial y tiene un toque especial con sabor español.
Un nuevo motor para el diseño de tensiones
El estudio de IA y automatización fue dirigido por Taek Soon Lee, director de Ingeniería Metabólica y de Vías en el JBEI, y Héctor García Martín, director de Ciencia de Datos y Modelado en el JBEI, ambos científicos de la División de EEB del Laboratorio Berkeley. Se propusieron acelerar uno de los pasos más laboriosos de la biología sintética: mejorar la producción microbiana mediante una serie de modificaciones genéticas en diferentes combinaciones de genes. Tradicionalmente, los científicos modifican unos pocos genes a la vez y prueban los resultados: un proceso minucioso, basado en la intuición, que puede tardar meses o incluso años en producir resultados significativos.
En cambio, los investigadores del Laboratorio Berkeley construyeron un sistema automatizado que utiliza robótica para crear y probar cientos de diseños genéticos en paralelo. Tras cada ronda, algoritmos de aprendizaje automático analizan los resultados para sugerir sistemáticamente el siguiente conjunto de diseños genéticos de cepas. El resultado es un sistema que avanza de 10 a 100 veces más rápido que los métodos convencionales.







Deja tu comentario
Tu dirección de correo electrónico no será publicada. Todos los campos son obligatorios